求助PDF
{"title":"Hidden histories in structure variations","authors":"Jun Lyu","doi":"10.1038/s41477-024-01888-5","DOIUrl":null,"url":null,"abstract":"","PeriodicalId":18904,"journal":{"name":"Nature Plants","volume":"11 1","pages":"7-7"},"PeriodicalIF":13.6000,"publicationDate":"2025-01-02","publicationTypes":"Journal Article","fieldsOfStudy":null,"isOpenAccess":false,"openAccessPdf":"","citationCount":"0","resultStr":null,"platform":"Semanticscholar","paperid":null,"PeriodicalName":"Nature Plants","FirstCategoryId":"99","ListUrlMain":"https://www.nature.com/articles/s41477-024-01888-5","RegionNum":1,"RegionCategory":"生物学","ArticlePicture":[],"TitleCN":null,"AbstractTextCN":null,"PMCID":null,"EPubDate":"","PubModel":"","JCR":"Q1","JCRName":"PLANT SCIENCES","Score":null,"Total":0}
引用次数: 0
引用
批量引用
结构变化中的隐藏历史
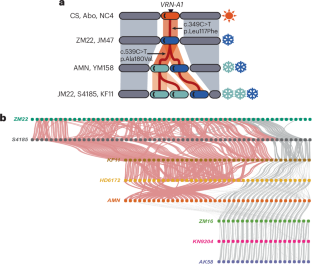
测序技术的进步已经使许多物种的全基因组可用,这提高了我们对自然变异和物种进化的理解(M. Schreiber et al.)。热纳Rev. 25, 563-577;2024)。在最近一期的《自然》杂志上,中国农业科学院作物科学研究所的焦承志及其同事报道了自20世纪50年代以来在中国不同时期发布的17个小麦品种的基因组组装。他们的分析将小麦基因组结构变异(SVs)与环境适应、迁徙和人类饮食偏好联系起来。这17个小麦品种代表了70年的育种历史,是根据它们对中国现代小麦育种和生产的贡献选出的。利用PacBio HiFi测序和Hi-C,研究人员获得了所有这些品种的染色体水平的高邻接组装。整合先前发表的四个基因组,他们进行了泛基因组分析,并确定了核心和不可或缺的基因家族。虽然当基因组数量(n)达到10个时,基因家族数量接近饱和,但即使在n = 21时,核苷酸结合富亮氨酸重复(NLR)基因也未达到饱和,这表明抗病基因具有更高的多样性,而这种多样性尚未在更大的种质中发现。
本文章由计算机程序翻译,如有差异,请以英文原文为准。