bioRxiv|突破蛋白语言模型的上下文限制:基于Bi-Mamba和PPI图的LC-PLM蛋白大模型
智药邦
2024-11-09 09:00
文章摘要
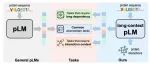
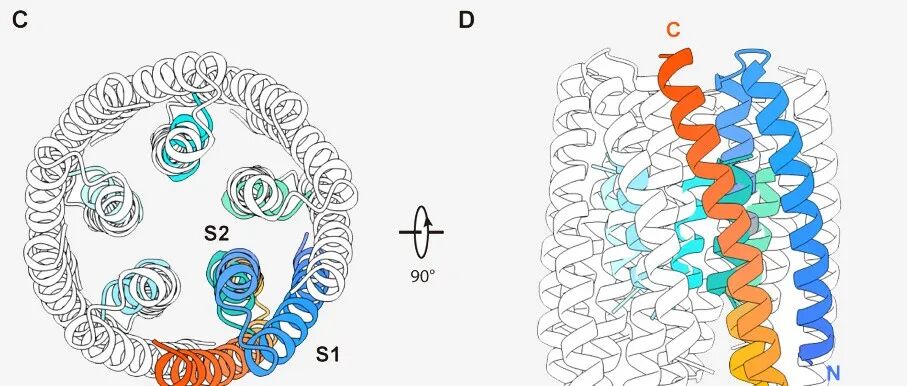
本文介绍了一种名为LC-PLM的新型蛋白质语言模型,旨在克服传统Transformer模型在处理长蛋白质序列和蛋白质相互作用时的局限性。LC-PLM基于BiMamba-S架构,通过选择性结构状态空间模型(SSM)提升长上下文处理能力,并采用双向共享投影层设计以捕捉氨基酸级别的细粒度信息。此外,LC-PLM的变体LC-PLM-G结合了蛋白质-蛋白质相互作用(PPI)图,进一步提升了蛋白质结构和功能预测任务的表现。实验结果显示,LC-PLM在长蛋白质序列建模和蛋白质结构预测任务中显著优于现有的Transformer模型,尤其是在处理蛋白质相互作用和复杂生物网络时表现出色。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。