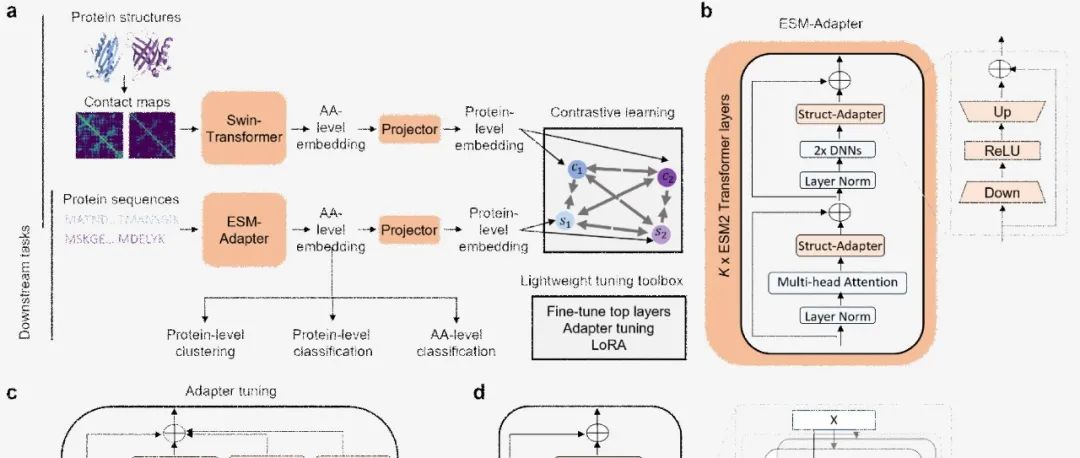
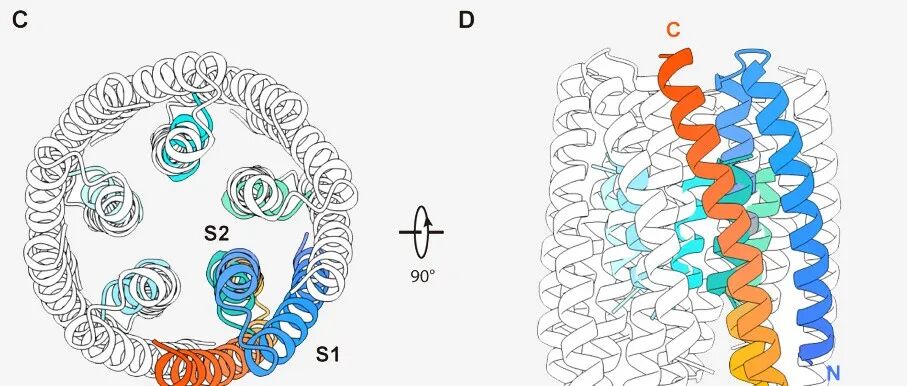
密苏里大学许东团队提出S-PLM模型,通过序列和结构对比学习的语言模型进行蛋白质预测
智药邦
2025-01-27 08:00
文章摘要
本文介绍了密苏里大学许东团队提出的S-PLM模型,这是一种通过序列和结构对比学习的蛋白质语言模型,旨在提高蛋白质预测的准确性。S-PLM模型通过集成Swin-Transformer和ESM2模型,有效地将蛋白质的3D结构信息融入到序列表示中,从而在蛋白质功能预测和结构分类任务中超越了现有方法。研究背景强调了蛋白质在生化过程中的重要性以及现有蛋白质语言模型在缺乏3D结构信息方面的局限性。研究目的是开发一种能够同时处理蛋白质序列和3D结构信息的模型,以提高预测性能。实验结果表明,S-PLM在多个蛋白质分类任务中均表现出色,特别是在结构感知方面,其序列表示能力显著优于仅依赖序列信息的模型。结论指出,S-PLM及其轻量级调优策略为蛋白质分析和预测任务提供了新的可能性,并有望通过进一步的数据增强和模型改进实现更广泛的应用。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。